5-methylcytosin
| 5-methylcytosin | |
|---|---|
 Strukturní vzorec | |
| Obecné | |
| Systematický název | 4-amino-5-methylpyrimidin-2-(1H)on |
| Sumární vzorec | C5H7N3O |
| Identifikace | |
| Registrační číslo CAS | 554-01-8 |
| EC-no (EINECS/ELINCS/NLP) | 209-058-3 |
| PubChem | 65040 |
| ChEBI | 27551 |
| SMILES | O=C1/N=C\C(=C(\N)N1)C |
| InChI | 1S/C5H7N3O/c1-3-2-7-5(9)8-4(3)6/h2H,1H3,(H3,6,7,8,9) |
| Vlastnosti | |
| Molární hmotnost | 125,13 g/mol |
| Teplota tání | 270 °C (543 K)[1] |
| Bezpečnost | |
| [1] | |
| H-věty | H317 H319[1] |
| P-věty | P261 P264+265 P272 P280 P302+352 P305+351+338 P321 P333+317 P337+317 P362+364 P501[1] |
Některá data mohou pocházet z datové položky. | |
5-Methylcytosin je organická sloučenina, methylovaná forma nukleové báze cytosinu, sloužící jako regulátor transkripce genů, i v několika dalších biologických dějích.[2] Při methylování cytosinu se posloupnost DNA (pořadí nukleových bází) nemění, ale může se měnit exprese takto upraveného genu. 5-Methylcytosin je součástí nukleosidu 5-methylcytidinu.
Objev[editovat | editovat zdroj]
W. G. Ruppel v roce 1898 při pokusu o izolaci bakteriálního toxinu zodpovědného za tuberkulózu z Mycobacterium tuberculosis získal novou nukleovou kyselinu, kterou nazval kyselina tuberkulinová.[3] Ukázalo se, že tato látka má neobvyklou strukturu, protože kromě thyminu, guaninu, a cytosinu, obsahuje také methylovaný nukleotid. V roce 1925, bylo detekováno malé množství methylovaného cytosinu jako produktu hydrolýzy kyseliny tuberkulinové kyselinou sírovou.[4][5] Vydaný článek byl napadán, protože určení produktu bylo založeno pouze na optikých vlastnostech krystalického pikrátu, a výsledky se nepodařilo zopakovat;[6] v roce 1948 ale Rollin Douglas Hotchkiss potvrdil jeho existenci při separaci nukleových kyselin z telecího brzlíku pomocí papírové chromatografie, kde detekoval methylovaný cytosin, látku odlišnou od běžného cytosinu i uracilu.[7]
Později byl methylcytosin nalezen také v mnoha molekulách RNA, kde ale jeho účel není znám.[8]
Funkce v organismech[editovat | editovat zdroj]
Funkce 5-methylcytosinu mohou být různé:[9]
- V bakteriích se 5-methylcytosin vyskytuje na řadě míst, často chrání DNA před štěpením restrikčními endonukleázami.
- U rostlin se objevuje v CpG, CpHpG a CpHpH (H = A, C nebo T).
- U hub a živočichů je 5-methylcytosin přítomen převážně v dinukleotidech. Většina eukaryot methyluje pouze malou část těchto míst, ale u obratlovců je methylováno 70-80 % CpG cytosinů. V buňkách savců se skupiny CpG na 5'-koncích genů označují jako CpG ostrovy.[10] 1 % veškeré savčí DNA je tvořeno 5mC.[11]
Zatímco samovolnou deaminací cytosinu vzniká uracil, který je rizpoznáván a odstraňován enzymy opravujícími DNA, tak deaminace 5-methylcytosinu vytváří thymin, přičemž tato změna cytosinu (C) na thymin (T) může vyvolat tranziční mutaci.[12]
Aktivní enzymatická deaminace cytosinu nebo 5-methylcytosinu cytosindeaminázami ze skupiny APOBEC mohou být výhodné pro mnohé buněčné procesy a pro vývoj organismu.[13] Vliv deaminace na 5-hydroxymethylcytosin není prozkoumán takto podrobně.
V laboratořích[editovat | editovat zdroj]
Deaminaci, odstranění skupiny NH2, 5-methylcytosinu na thymin lze provést například kyselinou dusitou; obdobně se provádí i deaminace cytosinu na uracil (U).
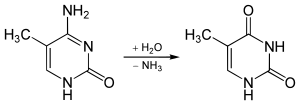
5-methylcytosin je odolný vůči deaminaci hydrogensiřičitany, které působí na nemethylovaný cytosin; této vlastnosti se využívá k analýze methylace cytosinu v DNA hydrogensiřičitanovým sekvencováním.[14]
[editovat | editovat zdroj]
5mC se na genomovou DNA navazují působením DNA methyltransferáz (DNMT). U člověka se vyskytuje 5 druhů DNMT, DNMT1, DNMT2, DNMT3A, DNMT3B, a DNMT3L, řasy a houby mají další 3 (DNMT4, DNMT5, DNMT6).[15] DNMT1 obsahuje replikační sekvence RFTS a domény CXXC, které katalyzují napojování 5mC. RFTS navádí DNMT1 na místa replikace DNA za spolupůsobení 5mC na dceřiných vláknech DNA, zatímco CXXC obsahuje zinkový prst, zajišťující de novo methylaci DNA.[16] DNMT1 je nejrozšířenější DNA methyltransferázou ve všech lidských tkáních.[17]
De novo methylaci zprostředkovávají hlavně DNMT3A a DNMT3B, za udržování 5mC značek při replikaci je zodpovědná DNMT1.[2] DNMT mohou interagovat mezi sebou a tím navyšovat kapacitu methylace; například 2 DNMT3L mohou vytvořit komplex se dvěma DNMT3A, čímž se zlepší interakce s DNA, což urychlí methylaci.[18]
Změny exprese DNMT vyvolávají odchylky v průběhu methylace. Nadměrná exprese způsobuje zvýšenou míru methylace, zatímco narušení funkce enzymu ji snižuje.[17]

Mechanismus methylace cytosinu začíná nukleofilním atakem cysteinového zbytku umístěného na PCQ motivu DNMT na uhlíku, který má být methylován. Následně S-adenosylmethionin poskytne methylovou skupinu a ta se naváže na uhlík v poloze 5. Zásaditá skupina v enzymu DNMT deprotonuje tento uhlík, čímž se odstraní přebývající atom vodíku a obnoví dvojná vazba mezi uhlíky 5 a 6, čímž vznikne 5-methylcytosin.[16]
Demethylace[editovat | editovat zdroj]
Po methylaci cytosinu na 5mC může opačný proces proběhnout několika způsoby. Pasivní demethylace DNA demethylation probíhá průběžně během replikace v důsledku absence údržby zprostředkované DNMT. Při aktivní demethylatci proběhne posloupnost oxidačních reakcí, jejímiž výsledky jsou postupně 5-hydroxymethylcytosin (5hmC), 5-formylcytosin (5fC), a 5-karboxylcytosin (5caC), přičemž poslední dvě uvedené molekuly jsou sestřiženy působením thymin-DNA glykosylázy (TDG), po které následuje opravná reakce za vzniku cytosinu.[2] TDG vytváří dvojnásobný nárust množství 5fC, aniž by došlo ke statisticky postřehnutelným změnám koncentrace 5hmC, což naznačuje, že 5mC se průběžně oxiduje nejméně dvakrát než dojde k úplné demethylaci.[19]
Oxidaci zajišťují dioxygenázy ze skupiny TET, které mohou pořeměnit 5mC, 5hmC, a 5fC na odpovídající oxidované molekuly; nejvyšší afinitu má ale enzym k 5mC a počáteční rychlostní konstanta přeměny 5hmC a 5fC pomoicí TET2 je 4,9-7,6krát nižší.[20]
TET potřebují železnatý kation jako kofaktor, a kyslík a α-ketoglutarát (α-KG) jako substráty; druhý uvedený substrát se tvoří z isocitrátu za katalýzy isocitrátdehydrogenázou (IDH).[21]
Nádorové buňky vyrábějí 2-hydroxyglutarát (2HG), který do těchto procesů vstupuje podobně jako α-KG, čímž kompetitivní inhibicí snižují aktivitu TET, a tak omezují přeměnu 5mC na 5hmC.[22]
Působení u lidí[editovat | editovat zdroj]
V nádorových buňkách[editovat | editovat zdroj]
V nádorových buňkách může být míra methylace DNA buď příliš vysoká (hypermethylace), nebo příliš nízká (hypomethylace).[23] CpG ostrůvky jsou de novo methylovány, což vede k nevhodné deaktivaci genů obvykle spojených s tlumením růstu nádorů.[24]
V nádorových tkáních jsou oproti zdravým vyšší koncentrace methyltransferáz DNMT1, DNMT3A, a především DNMT3B, přičemž se všechny podílejí na zvýšených hladinách 5mC.[17] Opakující se sekvence genomu jsou zde často hypomethylované, takže dochází k expresi jinak potlačovaných genů; intenzita této odchylky často odpovídá postupu nádoru.[23] Předpokládá se, že existuje spojení mezi hypermethylací a hypomethylací; nadměrná aktivita methyltransferáz vytvářející nadměrná množství de novo 5mC jsou vyrovnávána demethylacemi, odstranění methylových skupin je ale neúčinné, což vede k rozšíření hypomethylace po celém genomu; může nastat i opačný stav, kdy nadměrná exprese hypomethylace vyvolává hypermethylaci v rámci celého genomu.[23] Cisplatina reaguje s 5mC.[25]
Odkazy[editovat | editovat zdroj]
Reference[editovat | editovat zdroj]
V tomto článku byl použit překlad textu z článku 5-Methylcytosine na anglické Wikipedii.
- ↑ a b c d https://pubchem.ncbi.nlm.nih.gov/compound/65040
- ↑ a b c Xiaoji Wu; Yi Zhang. TET-mediated active DNA demethylation: mechanism, function and beyond. Nature Reviews Genetics. 2017-05-30, s. 517–534. ISSN 1471-0056. DOI 10.1038/nrg.2017.33. PMID 28555658.
- ↑ A. P. Matthews. Physiological Chemistry. [s.l.]: Williams & Wilkins, 2012. ISBN 978-1130145373. S. 167.
- ↑ T. B. Johnson; R. D. Coghill. The discovery of 5-methyl-cytosine in tuberculinic acid, the nucleic acid of the Tubercle bacillus. Journal of the American Chemical Society. 1925, s. 2838-2844. DOI 10.1021/ja01688a030.
- ↑ Grosjean H (2009). https://www.ncbi.nlm.nih.gov/books/NBK6489/#A79016 Nucleic Acids Are Not Boring Long Polymers of Only Four Types of Nucleotides: A Guided Tour] Landes Bioscience
- ↑ E. Vischer; S. Zamenhof; E. Chargaff. Microbial nucleic acids: the desoxypentose nucleic acids of avian tubercle bacilli and yeast. Journal of Biological Chemistry. 1949, s. 429-438. DOI 10.1016/S0021-9258(18)57100-3. PMID 18107446.
- ↑ R. D. Hotchkiss. The quantitative separation of purines, pyrimidines and nucleosides by paper chromatography. Journal of Biological Chemistry. 1948, s. 315-332. DOI 10.1016/S0021-9258(18)57261-6. PMID 18873306.
- ↑ J. E. Squires, H. R. Patel, M. Nousch, T. Sibbritt, D. T. Humphreys, B. J. Parker, C. M. Suter, T. Preiss. Widespread occurrence of 5-methylcytosine in human coding and non-coding RNA. Nucleic Acids Research. 2012, s. 5023-5033. DOI 10.1093/nar/gks144. PMID 22344696.
- ↑ V. Colot; J. L. Rossignol. Eukaryotic DNA methylation as an evolutionary device. BioEssays. 1999, s. 402-411. DOI 10.1002/(SICI)1521-1878(199905)21:5<402::AID-BIES7>3.0.CO;2-B. PMID 10376011.
- ↑ Adrian P. Bird. CpG-rich islands and the function of DNA methylation. Nature. 1986, s. 209-213. ISSN 0028-0836. DOI 10.1038/321209a0. PMID 2423876. Bibcode 1986Natur.321..209B.
- ↑ M. Ehrlich; R. Y. Wang. 5-Methylcytosine in eukaryotic DNA. Science. 1981-06-19, s. 1350-1357. ISSN 0036-8075. DOI 10.1126/science.6262918. PMID 6262918. Bibcode 1981Sci...212.1350E.
- ↑ A. Sassa; Y. Kanemaru; N. Kamoshita; M. Honma. Mutagenic consequences of cytosine alterations site-specifically embedded in the human genome. Genes and Environment. 2016, s. 17. DOI 10.1186/s41021-016-0045-9. PMID 27588157. Bibcode 2016GeneE..38...17S.
- ↑ R. Chahwan; S. N. Wontakal; S. Roa. Crosstalk between genetic and epigenetic information through cytosine deamination. Trends in Genetics. 2010, s. 443-448. DOI 10.1016/j.tig.2010.07.005. PMID 20800313.
- ↑ S. J. Clark; J. Harrison; C. L. Paul; M. Frommer. High sensitivity mapping of methylated cytosines. Nucleic Acids Research. 1994, s. 2990–2997. DOI 10.1093/nar/22.15.2990. PMID 8065911.
- ↑ Loïc Ponger; Wen-Hsiung Li. Evolutionary Diversification of DNA Methyltransferases in Eukaryotic Genomes. Molecular Biology and Evolution. 2005, s. 1119-1128. Dostupné online. ISSN 0737-4038. DOI 10.1093/molbev/msi098. PMID 15689527.
- ↑ a b Frank Lyko. The DNA methyltransferase family: a versatile toolkit for epigenetic regulation. Nature Reviews Genetics. 2018, s. 81-92. ISSN 1471-0064. DOI 10.1038/nrg.2017.80. PMID 29033456.
- ↑ a b c K. D. Robertson; E. Uzvolgyi; G. Liang; C. Talmadge; J. Sumegi; F. A. Gonzales; P. A. Jones. The human DNA methyltransferases (DNMTs) 1, 3a and 3b: coordinate mRNA expression in normal tissues and overexpression in tumors. Nucleic Acids Research. 1999, s. 2291-2298. ISSN 0305-1048. DOI 10.1093/nar/27.11.2291. PMID 10325416.
- ↑ Da Jia; Renata Z. Jurkowska; Xing Zhang; Albert Jeltsch; Xiaodong Cheng. Structure of Dnmt3a bound to Dnmt3L suggests a model for de novo DNA methylation. Nature. 2007, s. 248-251. ISSN 1476-4687. DOI 10.1038/nature06146. PMID 17713477. Bibcode 2007Natur.449..248J.
- ↑ Chun-Xiao Song, Keith E. Szulwach, Qing Dai, Ye Fu, Shi-Qing Mao, Li Lin, Craig Street, Yujing Li, Mickael Poidevin, Hao Wu, Juan Gao. Genome-wide profiling of 5-formylcytosine reveals its roles in epigenetic priming. Cell. 2013-04-25, s. 678-691. ISSN 1097-4172. DOI 10.1016/j.cell.2013.04.001. PMID 23602153.
- ↑ Shinsuke Ito, Li Shen, Qing Dai, Susan C. Wu, Leonard B. Collins, James A. Swenberg, Chuan He, Yi Zhang. Tet Proteins Can Convert 5-Methylcytosine to 5-Formylcytosine and 5-Carboxylcytosine. Science. 2011-09-02, s. 1300-1303. ISSN 0036-8075. DOI 21778364. Bibcode 2011Sci...333.1300I.
- ↑ Xingyu Lu; Boxuan Simen Zhao; Chuan He. TET Family Proteins: Oxidation Activity, Interacting Molecules, and Functions in Diseases. Chemical Reviews. 2015-02-12, s. 2225-2239. ISSN 0009-2665. DOI 10.1021/cr500470n. PMID 25675246.
- ↑ Wei Xu, Hui Yang, Ying Liu, Ying Yang, Ping Wang, Se-Hee Kim, Shinsuke Ito, Chen Yang, Pu Wang, Meng-Tao Xiao, Li-xia Liu. Oncometabolite 2-Hydroxyglutarate Is a Competitive Inhibitor of α-Ketoglutarate-Dependent Dioxygenases. Cancer Cell. 2011-01-18, s. 17-30. ISSN 1535-6108. DOI 10.1016/j.ccr.2010.12.014. PMID 21251613.
- ↑ a b c Melanie Ehrlich. DNA hypomethylation in cancer cells. Epigenomics. 2009, s. 239-259. ISSN 1750-1911. DOI 10.2217/epi.09.33. PMID 20495664.
- ↑ Peter A. Jones. DNA Methylation Errors and Cancer. Cancer Research. 1996, s. 2463-2467. Dostupné online. ISSN 0008-5472. PMID 8653676.
- ↑ Annika Menke; Romeo C. A. Dubini; Peter Mayer; Petra Rovó; Lena Daumann. Formation of Cisplatin Adducts with the Epigenetically-relevant Nucleobase 5-Methylcytosine. European Journal of Inorganic Chemistry. 2020-10-23, s. 30-36. ISSN 1434-1948. DOI 10.1002/ejic.202000898.
Externí odkazy[editovat | editovat zdroj]
 Obrázky, zvuky či videa k tématu 5-methylcytosin na Wikimedia Commons
Obrázky, zvuky či videa k tématu 5-methylcytosin na Wikimedia Commons
Literatura[editovat | editovat zdroj]
- Anthony J. F. Griffiths. An Introduction to genetic analysis. San Francisco: W. H. Freeman, 1999. ISBN 0-7167-3520-2. Kapitola Chapter 15: Gene Mutation.

