Genetický kód: Porovnání verzí
Robot: Opravuji 0 zdrojů a označuji 1 zdrojů jako nefunkční) #IABot (v2.0.1 |
Odebrání cizojazyčných pasáží. |
||
| Řádek 429: | Řádek 429: | ||
Degenerovanost genetického kódu a z ní plynoucí existence tichých [[mutace|mutací]] značně zvyšuje toleranci substitučních mutací v degenerovaných kodonech. Např. kodony kódující alanin (GCG, GCA, GCU, GCC) mohou po libosti mutovat na své třetí pozici, aniž by došlo k záměně [[Aminokyselina|aminokyseliny]], kterou kódují. Naproti tomu [[aminokyselina]] [[histidin]] je kódována pouze dvěma kodony, takže bez změny [[Aminokyselina|aminokyseliny]] je pouze jedna z možných tří mutací na třetí pozici. |
Degenerovanost genetického kódu a z ní plynoucí existence tichých [[mutace|mutací]] značně zvyšuje toleranci substitučních mutací v degenerovaných kodonech. Např. kodony kódující alanin (GCG, GCA, GCU, GCC) mohou po libosti mutovat na své třetí pozici, aniž by došlo k záměně [[Aminokyselina|aminokyseliny]], kterou kódují. Naproti tomu [[aminokyselina]] [[histidin]] je kódována pouze dvěma kodony, takže bez změny [[Aminokyselina|aminokyseliny]] je pouze jedna z možných tří mutací na třetí pozici. |
||
<!--The codons attempt to ensure that minor errors in the genetic code either only causes a silent mutation or an error that would not affect the amino acid's [[hydrophilic]]/[[hydrophobic]] property, eg. a codon of XUX (where X = any nucleotide) tends to code for hydrophobic amino acids. However, the genetic code is not perfect as minor changes such as the single-base substitution in [[sickle cell anaemia]] is disasterous. The hydrophilic [[glutamate]] (Glu) is substituted by the hydrophobic [[valine]] (Val) which reduces the solubility of ß-globin. This causes haemoglobin to form linear polymers linked by the hydrophobic interaction between the valine groups causing sickle cell deformation of erythrocytes. |
|||
These variable codes for amino acids are possible because of modified bases in the first base of the [[anticodon]], and the basepair formed is called a [[wobble base pair]]. The modified bases include [[inosine]] and the U-G basepair. |
|||
Only two amino acids are specified by a single codon; one of these is the amino-acid [[methionine]], specified by the codon AUG, which also specifies the start of transcription; the other is [[tryptophan]], specified by the codon UGG. |
|||
=== Phase or reading frame of a sequence === |
|||
Note that a „codon“ is entirely defined by your starting position. For example, the string GGGAAACCC, if read from the first position, contains the codons GGG, AAA and CCC. If read from the second position, it contains the codons GGA and AAC (partial codons being ignored). If read starting from the third position, GAA and ACC. Every DNA sequence can thus be read in three '''reading frames''', each of which will produce a radically different amino acid sequence (in our example, Gly-Lys-Pro, Gly-Asp, and Glu-Thr, respectively). The actual frame a protein sequence is translated in is defined by a '''start codon''', usually the first occurrence of AUG in the RNA sequence. Mutations that disrupt the reading frame (i.e. insertions or deletions of one or two nucleotide bases) severely impair the function of a protein and are thus exceedingly rare in protein-coding sequences, since they do not often survive [[purifying selection]]. |
|||
==Origin of the genetic code== |
|||
Numerous variations of the standard genetic code are found in [[mitochondrion|mitochondria]], which are energy-producing [[organelles]]. [[Ciliate]] [[protozoa]] also have some variation in the genetic code: UAG and often UAA code for Glutamine (a variant also found in some [[green alga]]e), or UGA codes for Cysteine. Another variant is found in some species of the [[yeast]] [[Candida (rod)|candida]], where CUG codes for Serine. In some species of [[bacterium|bacteria]] and [[archaea]], a few non-standard amino acids are substituted for standard stop codons; UGA can code for [[selenocysteine]] and UAG can code for [[pyrrolysine]]. There may be other non-standard interpretations that are not known. |
|||
Despite these variations, the genetic codes used by all known forms of life on Earth are very similar. Since there are many possible genetic codes that are thought to have similar utility to the one used by Earth life, the theory of [[evolution]] suggests that the genetic code was established very early in the history of life. |
|||
One can ask the question: is the genetic code completely random, just one set of codon-amino acid correspondences that happened to establish itself and be „frozen in“ early in evolution, although ''functionally'' any other of the near-infinite set of possible transcription tables would have done just as well? Already a cursory look at the table shows patterns that suggest that this is not the case. |
|||
Recent [[aptamer]] experiments have shown that amino acids have indeed a selective chemical affinity for the base triplets that code for them.{{fn|1}} This suggests that the current, complex transcription mechanism involving [[tRNA]] and associated enzymes is a later development, and that originally, protein sequences were directly templated on base sequences. Also, evidence has been found that originally the number of different amino acids used may have been considerably smaller than today.{{fn|2}} |
|||
== Reference == |
|||
There are several books available online that go into great detail on this topic. They are available through the [http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=Books NCBI Bookshelf], maintained by the [[National_Institutes_of_Health|United States National Institutes of Health]]. In particular the following books would be useful to consult: |
|||
* Griffiths, Anthony J.F.; Miller, Jeffrey H.; Suzuki, David T.; Lewontin, Richard C.; Gelbart, William M. (1999). [http://www.ncbi.nlm.nih.gov/books/bv.fcgi?call=bv.View..ShowTOC&rid=iga.TOC ''Introduction to Genetic Analysis'' (7th ed.)]. New York: W. H. Freeman & Co. ISBN 0-7167-3771-X |
|||
* Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter. (2002). [http://www.ncbi.nlm.nih.gov/books/bv.fcgi?call=bv.View..ShowTOC&rid=mboc4.TOC&depth=2 ''Molecular Biology of the Cell'' (4th ed.)]. New York: Garland Publishing. ISBN 0-8153-3218-1 |
|||
* Lodish, Harvey; Berk, Arnold; Zipursky, S. Lawrence; Matsudaira, Paul; Baltimore, David; Darnell, James E. (1999). [http://www.ncbi.nlm.nih.gov/books/bv.fcgi?call=bv.View..ShowTOC&rid=mcb.TOC ''Molecular Cell Biology'' (4th ed.)]. New York: W. H. Freeman & Co. ISBN 0-7167-3706-X |
|||
o--> |
|||
== Reference == |
== Reference == |
||
| Řádek 461: | Řádek 434: | ||
== Literatura == |
== Literatura == |
||
* Knight, R.D. and Landweber, L.F. (1998). [http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=pubmed&dopt=Abstract&list_uids=9751648 Rhyme or reason: RNA-arginine interactions and the genetic code.] ''Chemistry & Biology'' '''5'''(9), R215-R220. [https://web.archive.org/web/20050512032249/http://bayes.colorado.edu/Papers/chmbio98.pdf PDF version of manuscript] |
* Knight, R.D. and Landweber, L.F. (1998). [http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=pubmed&dopt=Abstract&list_uids=9751648 Rhyme or reason: RNA-arginine interactions and the genetic code.] ''Chemistry & Biology'' '''5'''(9), R215-R220. [https://web.archive.org/web/20050512032249/http://bayes.colorado.edu/Papers/chmbio98.pdf PDF version of manuscript] |
||
* Brooks, Dawn J.; Fresco, Jacques R.; Lesk, Arthur M.; and Singh, Mona. (2002). [https://web.archive.org/web/20041213094516/http://mbe.oupjournals.org/cgi/content/full/19/10/1645 Evolution of Amino Acid Frequencies in Proteins Over Deep Time: Inferred Order of Introduction of Amino Acids into the Genetic Code]. ''Molecular Biology and Evolution'' '''19''', 1645-1655. |
* Brooks, Dawn J.; Fresco, Jacques R.; Lesk, Arthur M.; and Singh, Mona. (2002). [https://web.archive.org/web/20041213094516/http://mbe.oupjournals.org/cgi/content/full/19/10/1645 Evolution of Amino Acid Frequencies in Proteins Over Deep Time: Inferred Order of Introduction of Amino Acids into the Genetic Code]. ''Molecular Biology and Evolution'' '''19''', 1645-1655. |
||
Verze z 1. 8. 2020, 22:14
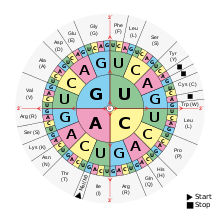
Genetický kód představuje soubor pravidel, podle kterých se genetická informace uložená v DNA (respektive RNA) převádí na primární strukturu bílkovin - tj. pořadí aminokyselin v řetězci. Genetický kód je univerzální - stejný u většiny živých organismů, pouze u několika málo skupin a mitochondrií se vyskytují drobné odchylky. Podoba genetického kódu společná většině živých organismů se nazývá standardní genetický kód.
Genová exprese

Genetická informace nesená organismem (jeho genom) je zapsaná v molekule DNA (s výjimkou některých nebuněčných organismů, u nichž tuto úlohu plní RNA). Každá funkční část (jednotka) DNA se nazývá gen. Každý gen se v procesu transkripce přepíše do odpovídající kratší molekuly mRNA, která slouží jako přenašeč informace od DNA k ribozómům - buněčným strukturám, na kterých probíhá translace (tvorba primární struktury bílkovin podle záznamu v mRNA). Pořadí aminokyselin se zde stanovuje tak, že ke každému kodonu (tripletu) se připojí tRNA s odpovídajícím antikodonem nesoucí aminokyselinu.
K jednotlivým kodonům tedy náleží odpovídající tRNA se specifickým antikodonem a specifickou aminokyselinou. Máme tedy 64 (43) možných kombinací, 64 odlišných kodonů. V následujících tabulkách je zaznamenán standardní genetický kód. Je z něj patrné, že genetický kód je degenerovaný - jedna aminokyselina může odpovídat většímu množství odlišných kodonů. Z tohoto důvodu také nelze podle vyrobené bílkoviny zrekonstruovat podobu genu, podle kterého byl vytvořen (viz Centrální dogma molekulární biologie).
Kodon
Kodon neboli triplet je označení tří za sebou jdoucích bází v mRNA. Určuje druh aminokyseliny. Ke každému kodonu existuje komplementární antikodon, což jsou vlastně tři za sebou jdoucí báze tRNA komplementární ke kodonu. Jednotlivá tRNA je specifická pro určitou aminokyselinu. Každá aminokyselina může být kódována více kodony, jeden kodon ale představuje pouze jednu aminokyselinu.[1]
Výjimečné postavení mají:
- Iniciační kodon - podle něj se pozná začátek genové sekvence nukleotidů v mRNA, začíná u něj translace, většinou AUG, výjimečně u prokaryot GUG, UUG, CUG
- Stop kodon - končí u něj proteosyntéza, UAA, UAG, UGA
Tabulka 1: RNA kodon → aminokyselina
| druhá báze | |||||
|---|---|---|---|---|---|
| U | C | A | G | ||
| první báze |
U |
UUU (Phe/F)Fenylalanin |
UCU (Ser/S)Serin |
UAU (Tyr/Y)Tyrosin |
UGU (Cys/C)Cystein |
| C |
CUU (Leu/L)Leucin |
CCU (Pro/P)Prolin |
CAU (His/H)Histidin |
CGU (Arg/R)Arginin | |
| A |
AUU (Ile/I)Isoleucin (Start)2 |
ACU (Thr/T)Threonin |
AAU (Asn/N)Asparagin |
AGU (Ser/S)Serin | |
| G |
GUU (Val/V)Valin |
GCU (Ala/A)Alanin |
GAU (Asp/D)Kyselina asparagová |
GGU (Gly/G)Glycin | |
1Kodon AUG slouží jako iniciační místo: první AUG v mRNA je místo, kde translace začíná; u eukaryot a archeí kóduje methionin, u bakterií (a z nich vzniklých organel - plastidů a mitochondrií) kóduje jako startovní kodon N-formylmethionin, dále v proteinovém řetězci pak standardní methionin
2Toto je startovní kodon pouze u některých prokaryot a v takovém případě pak kóduje methionin[2]
3U některých zelených řas, nálevníků, afelidií a diplomonád se vyvinul nekanonický kód, při kterém UAG a UAA translaci neukončují a namísto toho kódují glutamin, podobně UGA kóduje u některých nálevníků cystein a u rodu Mycoplasma tryptofan[3][4][5][6][7]
4U některých druhů archeí a baktérií je při biosyntéze enzymů pro metabolismus metanu normální Stop-funkce kodonu UAG modifikována přítomností zvláštní genové sekvence mRNA, umožňující navázat pyrolysinovou tRNA a zabudovat do proteinu pyrolysin (Pyl/O).
5Při biosyntéze některých proteinů archeí, baktérií i eukaryot je normální Stop-funkce kodonu UGA díky zvláštní genové sekvenci mRNA ignorována, což umožňuje navázat selenosysteinovou tRNA vzniklou selenizací serinové tRNA a zabudovat do proteinu selenocystein (Sec/U).
6V plastidech obrněnek Lepidodinium chlorophorum byla objevena odchylka kódu, při které kodon AUA kóduje methionin (Met/M)[8]
7U rodu Candida byla objevena odchylka kódu, při které kodon CUG kóduje serin (Ser/S)[9]
Mnoho výjimek, v tabulce nevyznačených, se vyskytuje v genetickém kódu mitochondrií a plastidů.[10]
Tabulka 2: aminokyselina → kodon(y)
| Ala | A | GCU, GCC, GCA, GCG | Lys | K | AAA, AAG |
| Arg | R | CGU, CGC, CGA, CGG, AGA, AGG | Met | M | AUG (AUU, GUG, UUG, CUG)2 (AUA)5 |
| Asn | N | AAU, AAC | Phe | F | UUU, UUC |
| Asp | D | GAU, GAC | Pro | P | CCU, CCC, CCA, CCG |
| Cys | C | UGU, UGC (UGA)1 | Pyl | O | UAG3 |
| Gln | Q | CAA, CAG (UAA, UAG)1 | Sec | U | UGA4 |
| Glu | E | GAA, GAG | Ser | S | UCU, UCC, UCA, UCG, AGU, AGC (CUG)7 |
| Gly | G | GGU, GGC, GGA, GGG | Thr | T | ACU, ACC, ACA, ACG |
| His | H | CAU, CAC | Trp | W | UGG (UGA)6 |
| Ile | I | AUU2, AUC, AUA5 | Tyr | Y | UAU, UAC |
| Leu | L | UUA, UUG2, CUU, CUC, CUA, CUG2,7 | Val | V | GUU, GUC, GUA, GUG2 |
| Start | AUG (AUU, GUG, UUG, CUG)2 | Stop | UAG1,3, UGA1,4,6, UAA1 |
1U některých zelených řas, nálevníků, afelidií a diplomonád se vyvinul nekanonický kód, při kterém UAG a UAA translaci neukončují a namísto toho kódují glutamin, podobně u některých nálevníků UGA kóduje cystein[3][4][5][6][7]
2U některých prokaryot je startovním kodonem také AUU, GUG, UUG či CUG, v takovém případě pak kóduje methionin[2]
3U některých druhů archeí a baktérií je při biosyntéze enzymů pro metabolismus metanu normální Stop-funkce kodonu UAG modifikována přítomností zvláštní genové sekvence mRNA, umožňující navázat pyrolysinovou tRNA a zabudovat do proteinu pyrolysin (Pyl/O).
4Při biosyntéze některých proteinů archeí, baktérií i eukaryot je normální Stop-funkce kodonu UGA díky zvláštní genové sekvenci mRNA ignorována, což umožňuje navázat selenosysteinovou tRNA vzniklou selenizací serinové tRNA a zabudovat do proteinu selenocystein (Sec/U).
5V plastidech obrněnek Lepidodinium chlorophorum byla objevena odchylka kódu, při které kodon AUA kóduje methionin (Met/M).[8]
6U rodu Mycoplasma byla objevena odchylka kódu, při které kodon UGA translaci neukončuje a namísto toho kóduje tryptofan (Trp/W)
7U rodu Candida byla objevena odchylka kódu, při které kodon CUG kóduje serin (Ser/S)[9]
Mnoho výjimek, v tabulce nevyznačených, se vyskytuje v genetickém kódu mitochondrií a plastidů.[10]
Marshall W. Nirenberg a jeho laboratoř (National Institutes of Health) první provedla pokusy, které ukázaly na závislost mezi kodony a aminokyselinami, které kódují. Har Gobind Khorana rozšířil Nirenbergovu práci a nalezl kódy pro aminokyseliny, které Nirenbergova metoda nalézt nemohla. Za svůj výzkum oba obdrželi Nobelovu cenu (1968).
Technické detaily
Stopkodony
Stopkodony, resp. terminační kodony jsou kodony ukončující translaci. Jsou tři: UAG, UGA a UAA a v anglické lit. jim přísluší následující nesystematická jména: UAG - amber (jantar), UGA - opal (opál), UAA - ochre (okr). Kodon amber pojmenovali jeho objevitelé Richard Epstein a Charles Steinberg po svém příteli Harrisi Bernsteinovi, jehož příjmení znamená v němčině "jantar". Zbývající dva kodóny byly pojmenovány vtipně opal a ochre pro zachování "barevného pojmenování".
U některých zelených řas, nálevníků, afelidií a diplomonád se vyvinul nekanonický kód, při kterém UAG a UAA translaci neukončují a namísto toho kódují glutamin, podobně UGA kóduje u některých nálevníků cystein a u rodu Mycoplasma tryptofan.[3][4][5][6][7]
U některých druhů archeí a baktérií je při biosyntéze enzymů pro metabolismus metanu normální Stop-funkce kodonu UAG modifikována přítomností zvláštní genové sekvence mRNA, umožňující navázat pyrolysinovou tRNA a zabudovat do proteinu pyrolysin. Při biosyntéze některých proteinů archeí, baktérií i eukaryot umožňuje podobný mechanismus ignorovat při translaci normální Stop-funkci kodonu UGA, navázat selenosysteinovou tRNA, vzniklou selenizací serinové tRNA, a zabudovat do proteinu selenocystein.
Startkodony
Startkodony, resp. iniciační kodony jsou místem, kde dochází k zahájení translace. Na rozdíl od stopkodonů ale přítomnost startkodónu sama o sobě nestačí, je ještě zapotřebí, aby se poblíž nacházela sekvence umožňující napojení mRNA na ribozóm.
Degenerovaný genetický kód
Genetický kód je degenerovaný, resp. redundantní, což znamená, že dva či více kodonů může kódovat jednu a tutéž aminokyselinu. Degenerované kodony se obvykle liší ve své třetí pozici, viz kodony GAA a GAG, které oba kódují glutamin. Tato degenerace genetického kódu umožňuje existenci tzv. tichých mutací.
Degenerovanost genetického kódu a z ní plynoucí existence tichých mutací značně zvyšuje toleranci substitučních mutací v degenerovaných kodonech. Např. kodony kódující alanin (GCG, GCA, GCU, GCC) mohou po libosti mutovat na své třetí pozici, aniž by došlo k záměně aminokyseliny, kterou kódují. Naproti tomu aminokyselina histidin je kódována pouze dvěma kodony, takže bez změny aminokyseliny je pouze jedna z možných tří mutací na třetí pozici.
Reference
- ↑ genetika.wz.cz. genetika.wz.cz [online]. [cit. 2008-01-10]. Dostupné v archivu pořízeném dne 2007-09-27.
- ↑ a b BLATTNER, F. R.; PLUNKETT, G.; BLOCH, C. A., et al. The complete genome sequence of Escherichia coli K-12. Science. 1997, roč. 277, čís. 5331, s. 1453-62. Dostupné online. ISSN 0036-8075. (anglicky)
- ↑ a b c COCQUYT, Ellen; GILE, Gillian H.; LELIAERT, Frederik, Heroen Verbruggen, Patrick J. Keeling, Olivier De Clerck. Complex phylogenetic distribution of a non-canonical genetic code in green algae. S. 1-24. BMC Evolutionary Biology [online]. 26. říjen 2010 [cit. 2010-10-29]. Svazek 10, čís. 327, s. 1-24. Dostupné online. PDF [1]. ISSN 1471-2148. DOI 10.1186/1471-2148-10-327. (anglicky)
- ↑ a b c HOFFMAN, David C.; ANDERSON, Richard C.; DUBOIS, Michelle L., Prescott David M. Macronuclear gene-sized molecules of hypotrichs. Nucleic Acids Research. 25. duben 1995, svazek 23, čís. 8, s. 1279-1283. Dostupné online [pdf]. PMID 7753617. (anglicky)
- ↑ a b c SCHNEIDER, Sigrid U.; LEIBLE, Michael B.; Xiao-Ping Yang. Strong homology between the small subunit of ribulose-1,5-bisphosphate carboxylase/oxygenase of two species of Acetabularia and the occurrence of unusual codon usage. Molecular and General Genetics. Únor 2001, svazek 218, čís. 3, s. 445-452. Abstrakt. Dostupné online. DOI 10.1007/BF00332408. PMID 2573818. (anglicky)[nedostupný zdroj]
- ↑ a b c KEELING, Patrick J.; DOOLITTLE, W. Ford. A non-canonical genetic code in an early diverging eukaryotic lineage. The European Molecular Biology Organization Journal. 1. květen 1996, svazek 15, čís. 9, s. 2285-2290. Dostupné online [pdf]. PMID 8641293. (anglicky)
- ↑ a b c KARPOV, Sergej A.; MICHAJLOV, Kirill V.; MIRZAEVA, Gulnara S., MIRABDULLAEV, Iskandar M.; MAMKAEVA, Kira A.; TITOVA, Nina N.; ALJOŠIN, Vladimir V. Obligately Phagotrophic Aphelids Turned out to Branch with the Earliest-diverging Fungi. S. 195-205. Protist [online]. 9. říjen 2012. Svazek 164, čís. 2, s. 195-205. Dostupné online. ISSN 1434-4610. DOI 10.1016/j.protis.2012.08.001. PMID 23058793. (anglicky)
- ↑ a b MATSUMOTO, Takuya; ISHIKAWA, Sohta A.; HASHIMOTO, Tetsuo, INAGAKI Yuji. A deviant genetic code in the green alga-derived plastid in the dinoflagellate Lepidodinium chlorophorum. S. 68-72. Molecular Phylogenetics and Evolution [online]. 22. duben 2011 [cit. 2011-05-30]. Svazek 60, čís. 1, s. 68-72. Dostupné online. ISSN 1055-7903. DOI 10.1016/j.ympev.2011.04.010. (anglicky)
- ↑ a b BUTLER, Geraldine; RASMUSSEN, Matthew D.; LIN, Michael F., Manuel A. S. Santos, Sharadha Sakthikumar, Carol A. Munro, Esther Rheinbay, Manfred Grabherr, Anja Forche, Jennifer L. Reedy, Ino Agrafioti, Martha B. Arnaud, Steven Bates, Alistair J. P. Brown, Sascha Brunke, Maria C. Costanzo, David A. Fitzpatrick, Piet W. J. de Groot, David Harris, Lois L. Hoyer, Bernhard Hube, Frans M. Klis, Chinnappa Kodira, Nicola Lennard, Mary E. Logue, Ronny Martin, Aaron M. Neiman, Elissavet Nikolaou, Michael A. Quail, Janet Quinn, Maria C. Santos, Florian F. Schmitzberger, Gavin Sherlock, Prachi Shah, Kevin A. T. Silverstein, Marek S. Skrzypek, David Soll, Rodney Staggs, Ian Stansfield, Michael P. H. Stumpf, Peter E. Sudbery, Thyagarajan Srikantha, Qiandong Zeng, Judith Berman, Matthew Berriman, Joseph Heitman, Neil A. R. Gow, Michael C. Lorenz, Bruce W. Birren, Manolis Kellis, Christina A. Cuomo. Evolution of pathogenicity and sexual reproduction in eight Candida genomes. Nature. 24. květen 2009, svazek 459, čís. 7247, s. 657–662. Dostupné online [html]. ISSN 0028-0836. DOI 10.1038/nature08064. (anglicky)
- ↑ a b ELZANOWSKI Andrzej, OSTELL Jim. The Genetic Codes, 7. červenec 2010. National Center for Biotechnology Information (NCBI), Bethesda, Maryland, USA.
Literatura
- Knight, R.D. and Landweber, L.F. (1998). Rhyme or reason: RNA-arginine interactions and the genetic code. Chemistry & Biology 5(9), R215-R220. PDF version of manuscript
- Brooks, Dawn J.; Fresco, Jacques R.; Lesk, Arthur M.; and Singh, Mona. (2002). Evolution of Amino Acid Frequencies in Proteins Over Deep Time: Inferred Order of Introduction of Amino Acids into the Genetic Code. Molecular Biology and Evolution 19, 1645-1655.
Externí odkazy
 Obrázky, zvuky či videa k tématu genetický kód na Wikimedia Commons
Obrázky, zvuky či videa k tématu genetický kód na Wikimedia Commons - Online DNA → Amino Acid Converter
